近日,太阳集团tyc234cc于彬教授团队在人工智能驱动药物发现研究领域取得重要进展,相关成果以“MTEGDRP: Interpretable Molecular Self-Attention Transformer and Equivariant Graph Neural Network Based on Multi-Omics Fusion for Drug Response Prediction in Cancer Cell Lines”为题发表在药物化学领域顶级期刊Journal of Medicinal Chemistry (JMC)。于彬教授为论文的唯一通讯作者,2023级研究生刘志涵为第一作者,太阳集团tyc234cc为第一完成单位。

随着人工智能和生物信息学的快速发展,肿瘤药物反应预测 (Cancer Drug Response Prediction, CDR) 已成为精准医学与计算药物学领域的重要研究方向。该研究聚焦于肿瘤细胞对抗癌药物的敏感性与耐药性差异,旨在通过系统建模药物分子特征与肿瘤多组学信息之间的复杂关系,为不同肿瘤个体筛选最优治疗方案。这一方向对于提升临床用药有效性、降低不良反应以及推动个性化治疗策略的制定具有重要意义。传统的药物反应研究方法主要依赖体外实验和临床试验,成本高、周期长,且难以全面刻画肿瘤分子层面的异质性。随着高通量测序技术的发展,基因组、转录组和表观遗传组等多组学数据不断积累,为系统解析肿瘤分子特征提供了数据基础。借助人工智能强大的特征学习和非线性建模能力,CDR 研究能够在大规模数据中自动挖掘影响药物反应的关键分子模式,从而实现对肿瘤药物敏感性的精准预测。然而,肿瘤药物反应预测仍面临若干关键挑战。一方面,多组学数据维度高、噪声大,特征冗余问题突出,另一方面,现有方法对药物分子结构信息的建模仍较为粗略,难以充分刻画其真实的空间几何特性。此外,多模态数据融合过程中对药物-肿瘤复杂交互关系的刻画仍有待加强。这些问题在一定程度上制约了模型预测性能和实际应用效果。因此,建立能够有效融合多组学信息与药物结构特征、并具备良好泛化能力和生物学解释性的预测模型,对于推动肿瘤药物反应预测方法向临床应用转化具有重要的研究价值和应用前景。
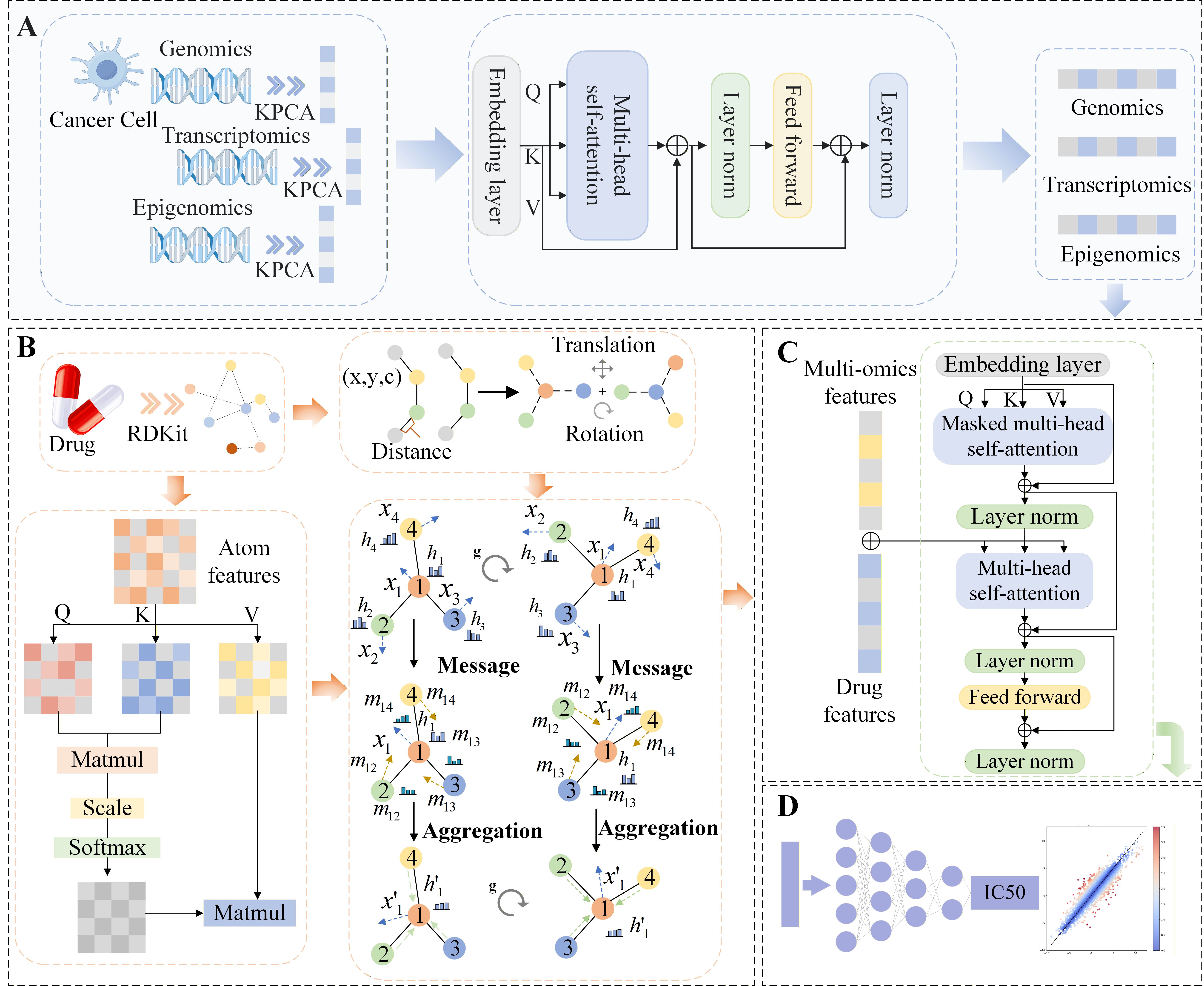
研究团队提出一种名为 MTEGDRP 的多组学融合深度学习框架。该模型创新性地整合了分子自注意力 Transformer 与等变图神经网络 (EGNN),旨在全面捕捉细胞系与药物间的复杂交互模式。具体而言,模型利用 Transformer 编码器对细胞系多组学特征进行深度表示学习,从而有效捕获不同组学特征之间的全局依赖关系。同时,模型首次在肿瘤药物反应预测任务中引入等变图神经网络,结合原子三维坐标与理化性质对药物分子的空间结构进行显式建模,从而弥补了传统方法在刻画药物几何信息方面的不足。此外,模型采用带有自注意力机制的 Transformer 解码器,深度解析药物与细胞系间的动态响应关系,实现多模态信息的有效对齐与融合。实验结果表明,MTEGDRP 在 GDSC2 数据集上的表现显著优于现有主流基线方法。在盲测实验中,该模型展现出强大的泛化能力,能够准确预测新药物和新细胞系的反应。消融研究进一步证实,多组学数据的整合以及药物空间信息的引入对提升预测精度起到了至关重要的作用。值得一提的是,该研究通过对药物分子权重和基因表达特征的视觉化分析,赋予了模型良好的生物学可解释性。可视化结果显示,模型能够精准识别出决定药物活性的核心功能基团 (如苯环、三氟甲基等),并捕捉到在药物反应中起关键作用的生物标志物。这一突破不仅为肿瘤药物反应机制的深入理解提供了新的研究视角,也在方法层面为揭示药物分子结构与肿瘤分子特征之间的作用规律奠定了基础,同时为精准医疗背景下的抗癌药物设计、药物重定位以及临床个性化用药决策提供了重要的理论依据和有效的计算辅助工具。
原文链接:https://doi.org/10.1021/acs.jmedchem.5c03438.
此外,该团队近一年来在AI药物发现领域取得三项重要科研成果,已发表论文:
PTET-DDI: Dual-channel drug-drug interaction prediction with a pre-trained language model and equivariant graph Transformer. ACS Synthetic Biology, 2026. (JCR 1区 IF=3.9) https://doi.org/10.1021/acssynbio.5c00722. 于彬教授、陈卓副教授为论文的通讯作者,研究生满孝锋、孙超并列第一作者。
MSN-DTA: A multi-scale node adaptive graph neural network for interpretable drug-target binding affinity prediction. Knowledge-Based Systems, 2025, 320: 113699. (JCR 1区 TOP IF=7.6). 于彬教授、于旭教授为论文的通讯作者,研究生王健鑫为第一作者。
BiVAE-CPI: An interpretable generative model using bilateral variational autoencoder for compound-protein interaction prediction. Journal of Chemical Information and Modeling, 2025, 65(17): 9313-9329. (JCR 1区TOP IF=5.3). 于彬教授、宋江宁教授为论文的通讯作者,研究生朱永鑫为第一作者。
以上研究成果得到了国家自然科学基金面上项目、山东省自然科学基金面上项目的资助。